摘要
稀有人参皂苷是人参药材及其加工产品体内发挥生物活性的主要物质基础,具有极高的应用价值和市场前景。由于稀有人参皂苷在天然人参植物中的含量极低,其高效制备策略成为人参研究领域的热点。通过结合现代生物转化技术与代谢工程方法,利用特定酶系及细胞工厂可实现高效、定向、绿色的稀有人参皂苷制备与合成。这些方法不仅提高了稀有人参皂苷的储备与化学多样性,还有效解决了传统化学方法中存在的环境友好性和副产物等问题。因此,该文综述了近年来关于稀有人参皂苷酶催化和代谢工程研究的进展,为打破行业技术壁垒、推动人参相关产业的高速发展提供参考和理论依据。
Abstract
Rare ginsenosides are the primary bioactive substances in ginseng and its processed products, possessing significant application value and market potential. However, due to their extremely low natural abundance in ginseng plants, efficient preparation strategies have become a focal point in ginseng research. By integrating modern biotransformation technologies with metabolic engineering approaches, the use of specific enzyme systems and microbial cell factories enables the efficient, targeted, and eco-friendly synthesis of rare ginsenosides. These methods not only enhance the reserves and chemical diversity of rare ginsenosides but also address environmental concerns and byproduct issues associated with traditional chemical methods. This review summarizes recent progress in enzymatic catalysis and metabolic engineering research on rare ginsenosides, providing insights and theoretical foundations to overcome industry technical barriers and promote the rapid development of ginseng-related industries.
Keywords
人参(Panax ginseng C. A. Mey.)被誉为“百草之王”,是我国的“名片中药”之一,其产业规模在中药行业中多年以来一直占据领先地位[1]。人参皂苷是人参属药用植物(人参、西洋参、三七等)的主要活性物质,也是《中国药典》中“人参”项下规定的指标性成分,对阐释人参药材的传统功效机制、指导临床合理用药、助推人参资源创新开发和保障人参产业的高质量可持续发展有着至关重要的作用[2]。目前,已从人参的不同药用部位(包括根、叶、果实和花蕾)中分离和鉴定得到超过 150 种人参皂苷[3]。根据其化学骨架和连接的糖基特点,人参皂苷主要分为 3 种类型:(1)达玛烷型(Dammarane,DA)皂苷,包括原人参二醇型(Protopanaxadiol,PPD)和原人参三醇型 (Protopanaxatriol,PPT)的四环三萜皂苷;(2)齐墩果酸型(Oleanolic acid,OA),以齐墩果酸为骨架的五环三萜人参皂苷;(3)奥克梯隆型(Ocotillol-type, OT)皂苷,C17 侧链中含有呋喃环的四环三萜皂苷[4-6]。根据人参皂苷的来源还可以将该类成分粗略地分为“原型人参皂苷”和“稀有人参皂苷”两种。其中,能够直接从人参自然资源中高效富集且含量较高的为原型人参皂苷,主要包括 Rg1、Re、Rf、 Rb1、Ro、Rc、Rb2 和 Rd,但这类成分普遍在胃肠道中的吸收较差,生物利用度较低[7]。稀有人参皂苷是从原型人参皂苷衍生的次级代谢产物,包括F2、 Rg3、Rh2、Compound K(C-K)、Compound O(C-O)、Compound Y(C-Y)、Rg2、Rh1 和 F1 等[8]。稀有人参皂苷的化学多样性较强,生物活性丰富,包括抗癌、抗炎、抗心血管疾病、抗糖尿病,以及对神经系统和肝脏的保护作用等,与原型人参皂苷相比,显示出更优越的体内代谢行为和药理作用效果,因此成为人参皂苷类物质新药开发的主要原料和研究对象[9]。人参中主要原型皂苷与稀有皂苷的相关信息见表1。
表1人参中主要原型皂苷与稀有皂苷的相关信息

续表1

续表1

然而,稀有人参皂苷在天然人参植物中含量极低,这对其开发和应用造成了相当大的限制[10]。因此,近年来稀有人参皂苷的制备已成为人参领域的研究热点。传统制备稀有人参皂苷的方法主要涉及热处理、酸水解、碱水解等策略[11]。然而,这些方法往往导致副产物过多、环境友好度低、反应剧烈且难以控制等问题。相比之下,生物转化法具有高效、绿色、反应条件温和等优点,是近10年以来稀有人参皂苷制备的主要技术手段[12]。生物转化方法包括酶法和微生物发酵法。尽管微生物发酵成本低、操作简单,但在生物安全性和遗传稳定性方面存在挑战[13]。酶法相比微生物表现出更高的特异性和转化效率,具有广阔的应用前景[14]。例如,以人参皂苷 Rg3 为主要成分的参一胶囊,就是通过酶转化的方式使 Rg3含量提高了 410倍,产量提高了 270倍,纯度达到 95% 以上。目前,参一胶囊已成功制备上市,并作为抗肿瘤辅助药物应用在临床一线[15]。
另外,随着基因组学、转录组学和蛋白质组学技术的蓬勃发展,以及生物合成策略的持续进步,为在微生物细胞中重建生物合成途径,以生产具有更复杂结构的下游产物铺平了道路[16]。与酶法制备稀有人参皂苷相比,基于模式细胞工厂的代谢工程能够从简单原料出发,定向合成既定结构的稀有人参皂苷,具有可持续性和成本优势[17]。但代谢工程技术的门槛较高,尤其是涉及工业生产时的产量和纯化问题是主要的挑战。而酶法虽然受限于成本和稳定性,但其工艺简单,转化效率高、特异性强,产品纯度高,与代谢工程取长补短可以极大程度的满足稀有皂苷产业化的需求。因此,本文归纳了近些年来人参皂苷生物转化酶和代谢调控研究的主要进展,从中挖掘出与稀有人参皂苷生物催化合成直接相关的糖苷酶与糖基转移酶,并剖析利用酿酒酵母等微生物细胞工厂生产稀有人参皂苷的实例。通过这些发现与总结,为稀有人参皂苷的高效、低成本、规模化制备提供技术支持和依据,为相关人参健康产品和新药的研发与产业化奠定理论基础。
1 稀有人参皂苷的构效关系——揭秘其潜在的生物活性密码
近年来,稀有人参皂苷的关注度居高不下,究其原因是其生物活性普遍强于原型人参皂苷。例如,经过蒸制的人参、三七和西洋参在治疗癌症、心血管疾病和认知障碍方面比生品表现出更好的疗效,因为蒸制过程可以显著提高稀有人参皂苷的含量[18]。现代药理学研究表明稀有人参皂苷生物活性的多样性与其独特的化学结构有关。影响稀有人参皂苷疗效的关键因素包括:糖分子的数量、糖键的连接方式、C-17侧链上的修饰和C20位的立体构型。
关于糖分子数量对稀有人参皂苷生物活性的影响最初在抗肿瘤研究中发现。例如,天然存在的人参皂苷Rb1和Rc几乎没有抗肿瘤作用,而稀有人参皂苷Rg3和Rh2则表现出强大的疗效。针对一些特定的肿瘤类型,随着人参皂苷中糖基数量的减少,其抗肿瘤效果逐步增强。其中,不含糖基的PPD(皂苷元)的作用最为显著,有望成为抗肿瘤治疗的候选药物[19]。因此,现在普遍认为增加糖基数量会削弱人参皂苷的疗效。其原因可能与人参皂苷的疏水特性有关,糖基的存在降低了整个分子的亲脂性,使其难以穿透细胞膜[20]。然而,在某些情况下,连接糖基较少的人参皂苷元反而显示出更弱的生物活性。例如,晚期糖基化终产物(AGE)是糖尿病相关血管并发症的重要标志物,人参皂苷元PPD和PPT对AGE 的抑制活性IC50值分别为451.45、449.23 μmol/L;相对而言,在不同位置分别连接 1 个糖基的人参皂苷 Rh2(C3位糖基)、Rh1(C6位糖基)和 C-K(C20位糖基)的 IC50 值分别为 3.38、8.42、10.85 µmol/L,远高于它们的苷元。这表明糖基的引入在稀有人参皂苷的某些药理作用中起到关键的作用[21]。
糖的连接位置是影响稀有人参皂苷生物活性的另一个因素。在抗肿瘤作用方面,大多数活性较好的化合物是糖基连接在 C3 位的 PPD 型皂苷,而非糖基连接在 C6 位的 PPT 型皂苷[22]。有研究者为了阐明这种构效差异开展了一系列实验,结果表明,C3位的糖基有助于人参皂苷在体内更长时间的循环,并增强其对肿瘤的主动靶向能力[23]。PPD 型人参皂苷与 PPT型相比在抗糖基化、抑制血管紧张素转换酶、抗雄激素非依赖型前列腺癌等领域也表现出更强的作用效果。通过应用分子模拟对接实验初步揭示了潜在的构效差异机制:PPT型皂苷C6 位的糖基增加了这些分子与靶蛋白之间的空间位阻,导致靶点亲和力显著低于PPD型皂苷[21,24-25]。
另外,达玛烷型皂苷 C17 位脂肪侧链上(C20-27)的结构修饰对其生物活性有着显著的影响。针对阿尔茨海默病的治疗,Zhang 等[26] 利用转基因秀丽隐杆线虫对17种人参皂苷进行活性筛选,研究发现人参皂苷 Rk3 的作用效果显著强于人参皂苷 Rh1,而二者唯一的区别就是 Rk3 在 C20 位的羟基脱水在 C20 和 C21 位之间形成一个新的双键。Ma 等[27] 发现,含有 C20-22 双键的稀有人参皂苷对 HL-60和 Hep-G2细胞系显示出强大的肿瘤细胞抑制作用,IC50 值分别为 10.32、24.33 μmol/L。有报道指出,人参皂苷插入生物膜中的取向受 C17侧链上的氧化还原程度和羟基位置的影响,在某些情况下,消除 C24-25位的双键可提高生物活性[28]。例如,通过还原Rg3中C24-25位的双键,得到了化学稳定的二氢人参皂苷 Rg3(2H-Rg3),其显示出比 Rg3 更显著的抗血小板聚集作用[29]。此外,将 C24-25位双键水合之后在 C-25 位置添加羟基可显著影响稀有人参皂苷的药理活性与生物利用度,尤其在抗肿瘤方面,一些 C25-OH 衍生物甚至比其原型化合物活性强几十倍,可以作为非常有发展前景的抗肿瘤候选药物[30]。本人课题组利用生物转化技术大量催化合成了系列 C25-OH 稀有皂苷衍生物,其中包括新化合物 4 个;对它们进行抗炎和心肌细胞保护的活性筛选,初步总结了构效关系,进一步证实了 C24-25 位双键水合对稀有人参皂苷活性的重要影响[31-34]。
最后,蒸制或酸水解引起的人参皂苷在 C20 位的脱糖基化通常会导致从 20(S)-型向 20(R)-型稀有人参皂苷的差向异构化,如20(R)-Rg3、20(R)-Rh2 和 20(R)-Rs3 等[35]。针对同一药理模型,这些差向异构体的生物活性往往差异较大,甚至会出现反向结果。拿人参皂苷 Rg3 和 Rh2 的抗肿瘤研究举例,针对不同的肿瘤细胞系和作用靶点,有的研究证明 S 构型活性更强,有的研究则表明 R 型活性更强[36-38]。一些研究者通过实验初步证明了,导致这种差异化结果的主要原因是跟目标靶点结合口袋的氨基酸残基排布和疏水性有关,但关于稀有皂苷 (S/R)差向异构体构效关系的研究目前没有得到具有规律性的结果 [39-41]。稀有人参皂苷的一般构效关系见图1。
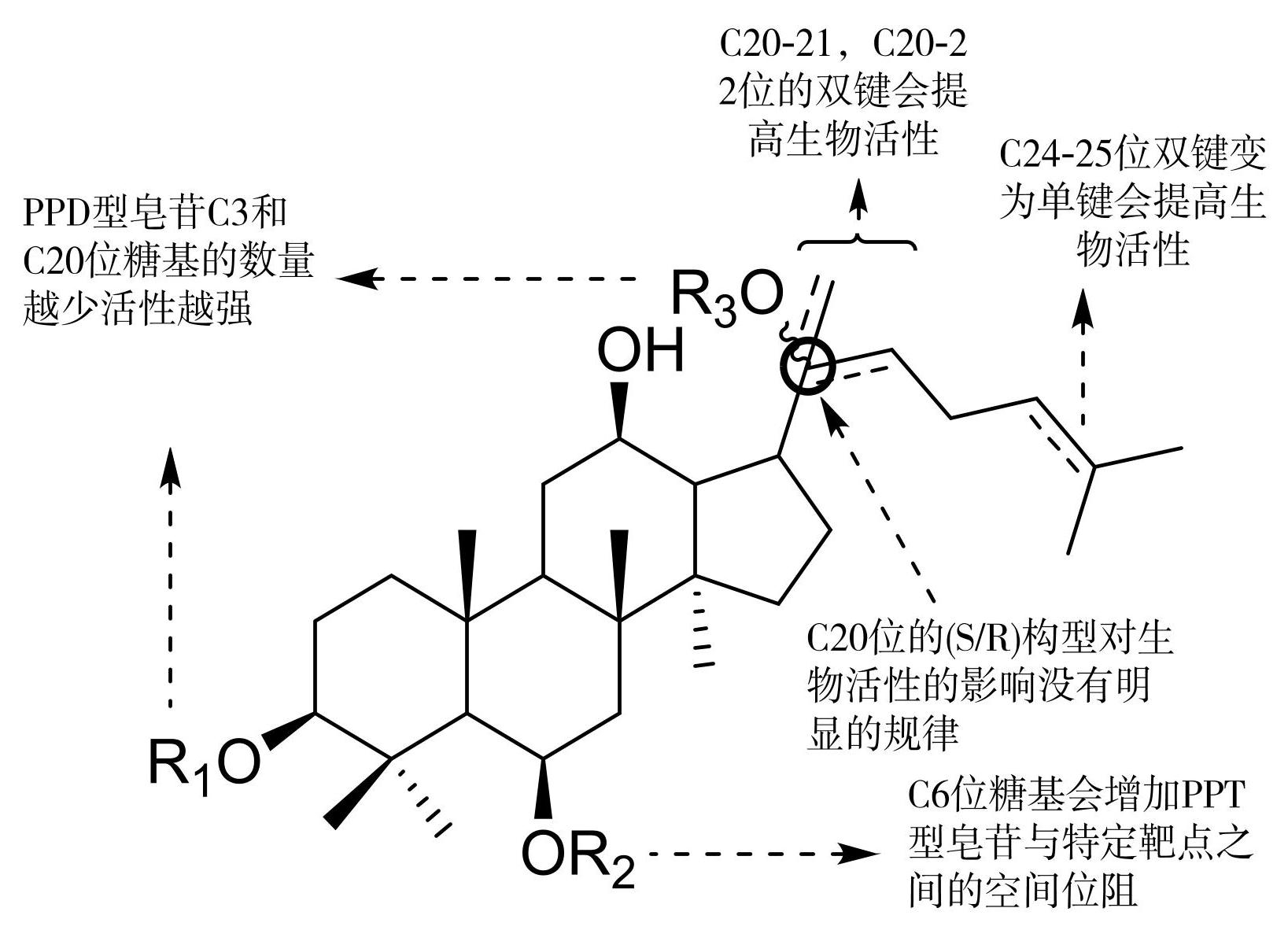
图1稀有人参皂苷的一般构效关系
2 稀有人参皂苷的酶催化制备研究进展
2.1 PPD型稀有人参皂苷的酶催化研究
人参皂苷Rg3是PPD型稀有人参皂苷的代表性化合物,也是第一个获批新药应用于临床辅助治疗癌症的人参皂苷类药物,因此对于 Rg3的制备一直以来都是稀有人参皂苷酶催化领域的热点[42]。目前可以作为底物进行人参皂苷Rg3制备的原型皂苷主要有 Rb1、Rb2、Rc和 Rb3,但无论选择哪种底物,均需先将其转化为 Rd,才能进一步生成 Rg3。为了生成人参皂苷Rd,Rb1、Rb2、Rc和Rb3需要分别将C20 外侧的糖基水解掉。然而,有别于人参皂苷 Rb1在 C20外侧的葡萄糖基,Rb2、Rc和 Rb3的 C20外侧位置分别为阿拉伯吡喃糖基、阿拉伯呋喃糖基和木糖基。而自然界中能够水解以上糖苷键的酶相对葡萄糖苷酶而言比较罕见,因此,人参皂苷Rb1是转化制备Rg3最为常用的底物,其转化途径为:Rb1→Rd→ Rg3[43]。通过筛选不同的酶,一些研究者成功实现了以Rb2和Rc为底物的20(S)-Rg3酶促转化。这些酶相对于一般的葡萄糖苷酶,热稳定性普遍较强,可以在 90℃下保持酶的催化活性;并且反应专属性较好,仅水解皂苷Rb2和Rc的C20末端糖苷键生成皂苷 Rd;通过进一步与葡萄糖苷酶联合应用,可以将 20(S)-Rg3 的摩尔转化率提高到 98.19%,显著降低了稀有人参皂苷20(S)-Rg3的制备成本[44-45]。
一般而言,C3位的糖苷键相对于C20位更难水解,这可能与C3所在的四环三萜骨架对糖苷酶进攻该位点造成空间位阻所致。随着 C3位外侧的糖基水解之后,C20 位糖基保持不变的难度就会越大。例如,在人参皂苷Rb1生物转化的过程中,如果出现了C3位糖苷键优先水解的情况,那么就会生成另外 2 种稀有皂苷 Gypenoside XVII(GypXVII)和 Gype‐ noside LXXV(GypLXXV)。一些研究者利用来自 Bifidobacterium dentium的重组糖苷酶,将Rb1的C3 位外侧葡萄糖基水解以生成 GypXVII,经过反应条件优化后,摩尔转化率可以达到100%[46]。
与人参皂苷 Rg3 相似,C-K 的酶催化过程也是从人参皂苷 Rb1、Rb2 和 Rc 开始,形成中间产物 Rd 之后,进一步水解完成的。区别在于,C-K 是由 Rd 水解掉C3位的两个葡萄糖基形成的,其主要的转化途径为(Rb1、Rb2、Rc)→Rd→F2→C-K,其难度要大于 Rd水解掉 C20位的糖基生成 Rg3,因此 C-K摩尔转化率一般为 60%~80%[51]。为进一步提高 C-K 的转化率,一些研究者通过蛋白质工程策略对已知的 β-糖苷酶进行定向进化,开发出耐高温的突变酶,可以通过转化Rb1、Rb2和Rc,实现100%的C-K 转化率,展现出极高的工业化生产潜力 [52-53]。从转化速率上看,Rb1 是 Rb2 和 Rc 的近 2 倍,这也说明了,葡萄糖苷键相对阿拉伯糖苷键更易于水解。为降低稀有人参皂苷 C-K 的生产成本,研究者们尝试利用人参总皂苷或者人参皂苷粗提物作为底物开展酶转化研究,取得了一些有意义的成果。例如, Jeong 等[54] 从 Aspergillus niger KACC 46495 中提取了高效胞外糖苷酶,通过羧甲基纤维素(CMC)诱导增强其糖苷水解活性之后,可以在 9 h 内将 6.0 g/L 的 PPD 型人参皂苷混合物转化为 2.8 g/L C-K,摩尔转化率达到80%,生产率为313 mg/(L·h)。
人参皂苷C-K的同分异构体Rh2也是一种极具药用价值的稀有皂苷,主要是通过水解人参皂苷 Rg3 的 C3 位外侧葡萄糖基或 F2 的 C20 位葡萄糖基生成的。然而,以人参皂苷 Rg3 或 F2 为底物制备 Rh2的成本较高,当以人参皂苷Rb1为底物时,目前发现的单一糖苷酶很难实现从Rb1→Rh2的专属反应,往往从 Rd 开始分化,一部分生成 F2 和 C-K,而另一部分生成Rg3和Rh2 [55]。应用组合酶法将能够水解特定位点和特定数量糖基的糖苷酶进行理性设计,可以实现人参皂苷 Rh2的定向转化。该过程主要分为两个阶段:首先,利用特定糖苷酶将Rb1和Rd等上游皂苷转化为中间产物 Rg3;然后通过加热使第一步的糖苷酶失活,再引入能够定向水解 Rg3 的 C3 位外侧糖基的糖苷酶,将 Rg3 完全转化为 Rh2。目前这种策略不仅仅应用在单体皂苷转化,而且在 PPD 型皂苷混合物转化生成 Rh2 方面也取得了良好的进展 [56-57]。
除了以上酶之外,还有一些糖苷酶具有复杂的底物选择范围或区域选择性,可以转化多种皂苷底物生成不同的稀有人参皂苷。这些酶主要分为3种情况:(1)针对不同皂苷的同一位点均可发生反应,说明该酶的区域选择性较好且底物选择范围较宽,可以针对不同生物活性的构效关系需求定向拓展化学多样性[58];(2)针对同一皂苷的不同位点均可发生反应,说明该酶的底物选择性较强,但区域选择性较差,可以用来做某种皂苷的结构修饰与衍生物制备[59];(3)针对不同皂苷的不同位点均可发生反应,说明该酶是一种广谱的糖苷水解酶,适用于对纯度要求不高的皂苷粗提物的生物催化[60-62]。PPD型稀有人参皂苷酶催化制备路线图见图2,PPD型稀有皂苷的酶催化相关参数见表2。
整体而言,PPD 型稀有人参皂苷的酶转化研究开展较为深入,几个主要的原型人参皂苷 C3 位和 C20 位的糖基水解酶均有报道,考虑到单体皂苷的成本较高,目前的研究以人参总皂苷或PPD型皂苷混合物为底物进行复杂体系的酶催化为主要趋势,以推进 PPD 型稀有人参皂苷的工业化开发和利用为主要目的。

图2PPD型稀有人参皂苷酶催化制备路线图
表2PPD型稀有皂苷的酶催化相关参数
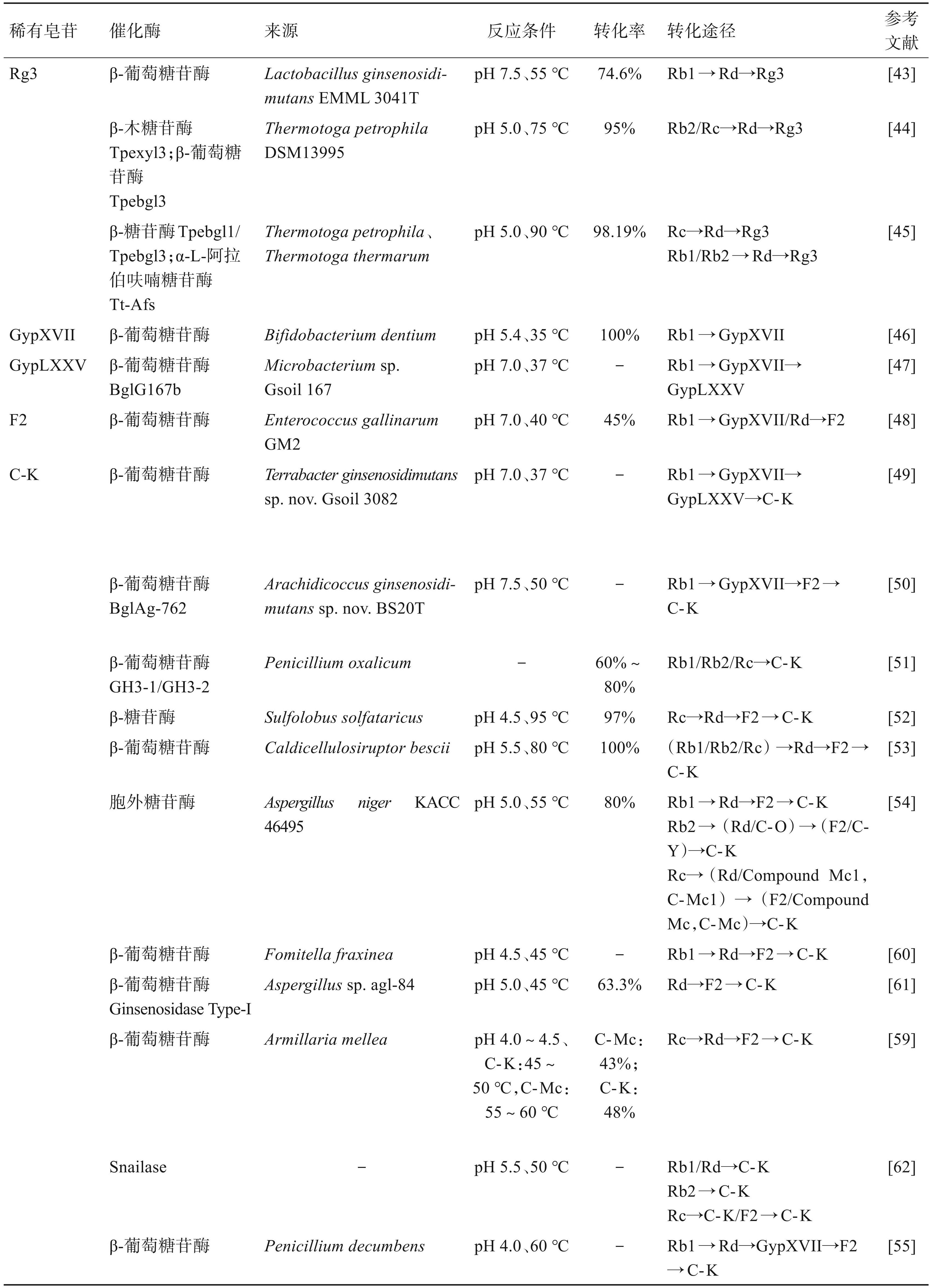
续表2

2.2 PPT型稀有人参皂苷的酶催化研究
PPD和PPT型皂苷在人参植物中的含量随季节性变化的趋势相似。有意思的是,人参根部富含 PPT和PPD型皂苷,而地上部分(如叶、茎和花)主要以 PPD 型皂苷为主[63]。尽管 PPT 型和 PPD 型皂苷在结构上高度相似,仅在糖苷键原子和羟基位置上有所区别,但是关于 PPT型皂苷的酶催化制备研究相对 PPD型较少,可能是因为 PPT型皂苷中新增的 C6位糖苷键更难被水解。
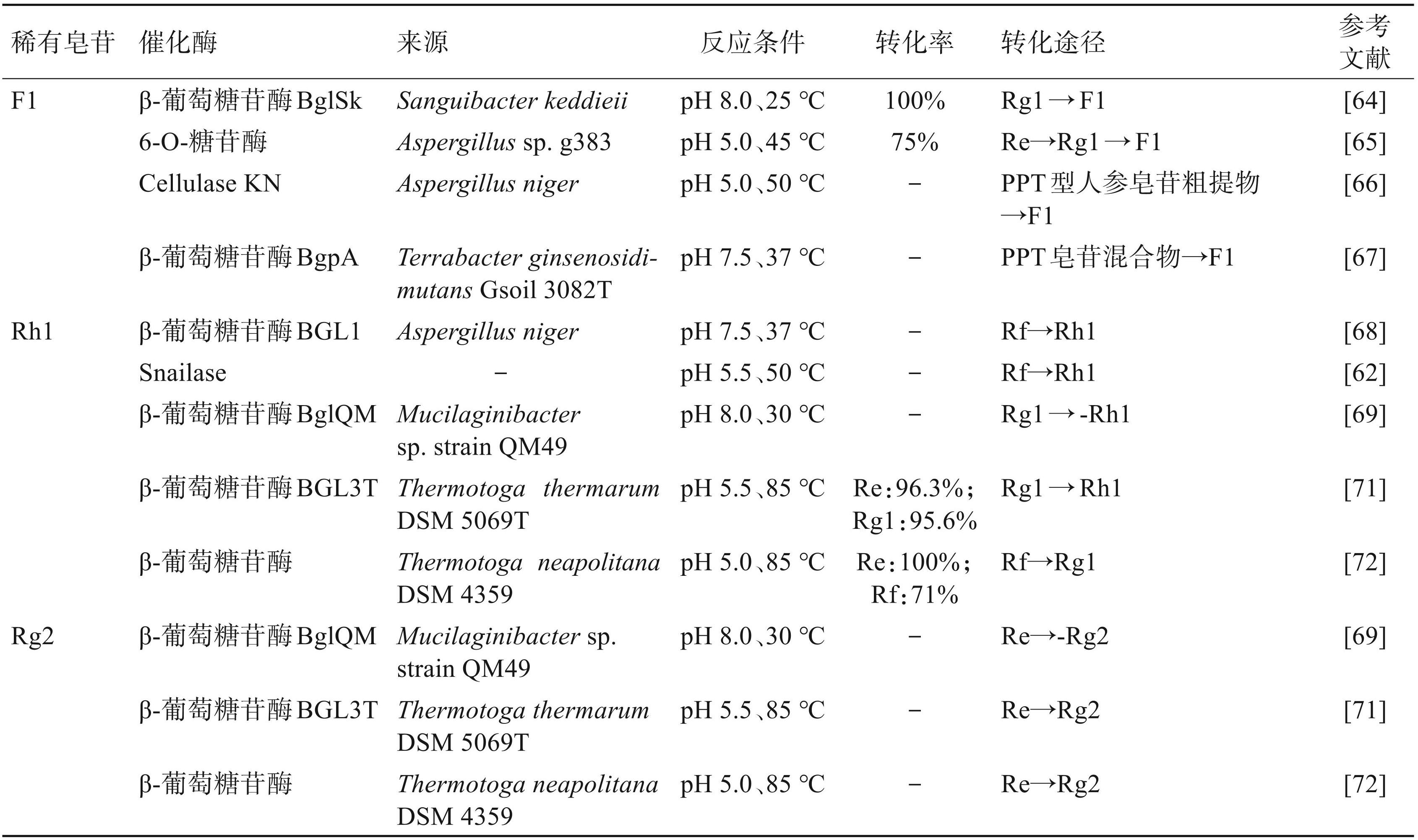
在PPT型皂苷中,人参皂苷Rg2、Rh1和F1是研究最为深入的稀有皂苷成分。其中 Rh1 和 F1 是同分异构体,具有不同的葡萄糖基连接位置:Rh1的葡萄糖基连接在 C6 位,而 F1 的葡萄糖基连接在 C20 位。因此,同时在C6和C20位连接有葡萄糖基的人参皂苷 Re和 Rg1就成为了制备稀有人参皂苷 F1或 Rh1 最常用的底物。从转化效果上看,利用 Rg1 转化生成 F1 只需要水解掉 C20 位的葡萄糖基,而从 Re 生成 F1 则需要同时水解掉 C6 位外侧的鼠李糖基和C20位的葡萄糖基,因此,以Rg1为底物催化制备 F1 的效率要高得多,通过优化之后可以达到 100% 的转化率[64]。反之,如果是以 Re 或者富含 Re 的PPT型总皂苷为底物催化制备F1,转化效率会显著下降,摩尔转化率很难超过75%[65-67]。
值得注意的是,当以人参皂苷 Re 为底物制备 Rh1的时候,虽然也会产生一定比例的 Rg2,但转化途径通常为:Re→Rg1→Rh1,而不是 Re→Rg2→ Rh1。因为,虽然人参皂苷 Rg2与 Rh1相比,仅仅是在C6位糖链外侧多了1个鼠李糖,但能够直接水解该鼠李糖生成 Rh1的糖苷酶较为少见,尤其是能够连续水解Re的C20位葡萄糖基和C6位末端的鼠李糖的糖苷酶罕有报道。相对而言,稀有人参皂苷 Rg2 连有两个糖基,且都在 C6 位,分别为近侧的葡萄糖基和外侧的鼠李糖基。与其他 PPT 型人参皂苷相比,人参皂苷 Re 的结构与 Rg2 最为相似,仅在 C20 位多了 1 个葡萄糖基。因此,关于稀有人参皂苷Rg2的制备研究均以人参皂苷Re为底物开展的,而人参皂苷Rh1的酶工程底物主要来源于人参皂苷 Rg1或Rf[62,68-69]。
一般而言,提高反应温度可以加快糖苷键水解的进程,尤其对于PPT型皂苷C6位这种难于水解的糖苷键,耐热酶的筛选更为重要。这些耐热糖苷酶主要来源于嗜热微生物,用于高效分解环境中的复杂碳水化合物,为微生物自身代谢提供能量。它们之所以能在高温下保持活性,主要归因于其分子结构的高度优化,包括更多的盐桥和氢键、更紧密的三级结构、疏水性核心的增强以及柔性区域的减少;此外,其亚基间的强结合、表面电荷分布的优化以及减少疏水表面暴露也大大提升了热稳定性[70]。目前,研究者们发现的耐热糖苷酶不仅可以在85℃ 的高温下实现 Re→Rg2(摩尔转化率 100%)和 Rg1 →Rh1(摩尔转化率 95.6%)的高效转化,甚至可以将Rf和Rg1转化为苷元PPT[摩尔转化率71%,生产率283 mg/(L·h)],进一步拓展了PPT型稀有皂苷的制备途径[71-72]。PPT型稀有人参皂苷酶催化制备路线图见图3,PPT 型稀有皂苷酶催化相关参数见表3。
当前关于稀有人参皂苷酶催化制备的研究主要集中在利用不同的糖苷酶水解 PPD 型皂苷 C3、 C20位和PPT型皂苷C6、C20位置的糖苷键,以改变糖链结构,获得具有更高生物活性的物质。尽管已有多种来源的糖苷酶可用于这一目的,但由于部分酶对目标位点的立体选择性较低或催化效率不足,使其在工业化生产中的实际应用受到限制。因此,挖掘天然存在的、具有多样化底物选择性和催化活性的糖苷酶,同时深入研究其在底物生物转化过程中的机制,对于推动稀有人参皂苷的生物修饰技术和合成工程酶的开发至关重要。
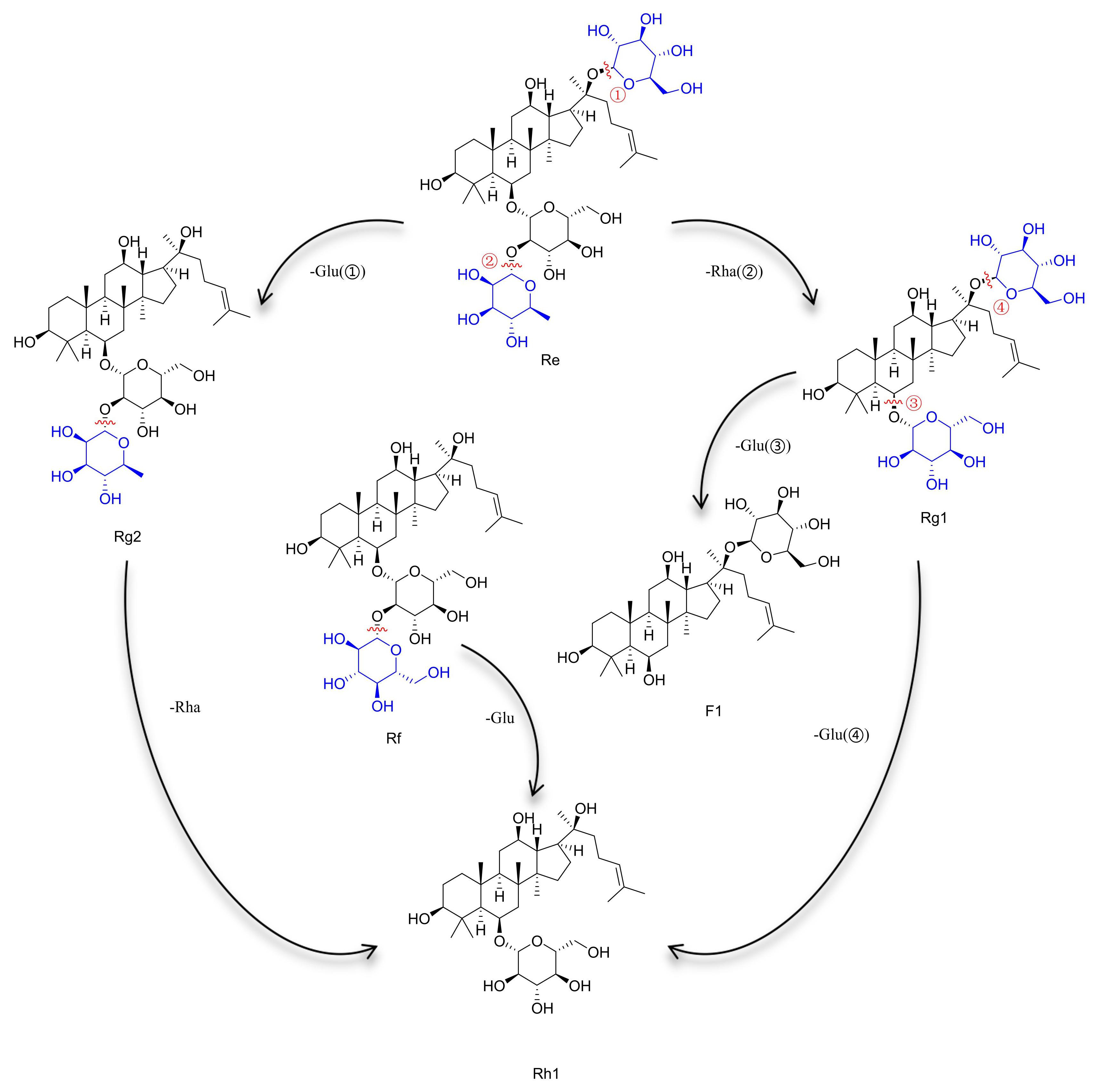
图3PPT型稀有人参皂苷酶催化制备路线图
表3PPT型稀有皂苷的酶催化相关参数
3 稀有人参皂苷的代谢工程研究进展
随着合成生物学技术的发展,利用微生物细胞工厂合成稀有人参皂苷已成为一种有效方法,并在近年来取得了显著进展,实现了包括C-K、Rh2、Rg3 和 F2 在内的多种稀有人参皂苷的从头合成。人参皂苷基本化学骨架生物合成的途径已经非常明晰,并且在其他文献中有详细的描述,其中达玛烷是稀有人参皂苷的主要骨架类型[73-75]。在达玛烷骨架形成后,通过细胞色素 P450(CYP450)和糖基转移酶(GT)的催化生成各种不同的稀有人参皂苷(见图4)。在这个过程中,CYP450 主要负责对骨架特定位点进行氧化修饰,为连接特定糖基奠定基础[76]; 然后再通过 GT 催化特定位点羟基的糖苷化,该过程是人参皂苷化学多样性的决定性步骤,也是稀有皂苷合成途径中的最后一步和关键限速反应[77]。为构建相关的生物合成工程菌,首先要打造能够高效合成稀有人参皂苷基本骨架的底盘。在这些生物合成底盘菌株中,酿酒酵母(Saccharomyces cerevisiae) 是1种稳定存在于单倍体和二倍体形式的单细胞真核生物,其遗传操作技术成熟且相对简单,是代谢工程研究中常用的模式生物[78-81]。同时,酿酒酵母也被认为是萜类化合物合成的理想底盘细胞,这主要归因于其甲戊二羟酸(MVA)途径与萜类化合物的合成途径具有高度相似性。因此,关于稀有人参皂苷代谢工程的研究主要是以酿酒酵母作为底盘菌株开展的。
3.1 PPD型稀有人参皂苷的代谢工程研究进展
3.1.1 苷元 PPD的代谢工程研究
PPD是 PPD型稀有人参皂苷的基本骨架,也是稀有人参皂苷生物合成发起的基石。在构建用于 PPD 生产的酿酒酵母工程菌株时,首先需要引入达玛烷二醇合成酶 (Dammarenediol Synthase,DDS)和原人参二醇合成酶(Protopanaxadiol Synthase,PPDS),以重构人参皂苷的生物合成途径。有研究表明,通过在酿酒酵母中异源表达来源于人参的 PgDDS 和 PgPPDS,以及拟南芥的 AtCPR1,可以实现 PPD 的生物合成。同时,过表达法尼基焦磷酸合成酶(FPS)、鲨烯合成酶 (SS)和鲨烯环氧化酶(SE)以增强前体供应,进而对 PgPPDS 进行密码子优化提高其酶活性,可以将 PPD的产量提升至 1 189 mg/L[82]。通过优化酿酒酵母中的 MVA 途径,例如对 3-羟基-3-甲基戊二酰辅酶A还原酶(HMGR)进行截短(tHMGR),可显著提高萜类化合物(如鲨烯)的产量[83-84]。采用类似的代谢工程策略,将来自不同植物的密码子优化的 DDS、PPDS、原人参三醇合成酶(Protopanaxadiol Synthase, PPTS)和AtCPR1引入过表达tHMGR、SS和 SE 的酿酒酵母菌株中,可同时合成 PPD 和 PPT[85]。然而,这些工程菌株中PPDS的代谢通量较低,严重限制了 PPD 的转化效率,这成为影响 PPD 产量的关键因素之一。通过去除PgPPDS的跨膜区域,并与AtCPR1融合后转入工程酵母菌株,可有效提高 PPD 的转化效率,使达玛烷二醇(DM)到 PPD 的转化率达到96.8%[86]。

图4稀有人参皂苷的常规生物合成途径
需要注意的是,在 DM 转化为 PPD 的过程中会产生活性氧自由基(ROS),其高积累会降低酵母细胞活力。尤其是当用乙醇替代葡萄糖作为乙酰辅酶 A(acetyl-CoA)供体时,ROS 的增加使酵母对乙醇更敏感,在乙醇和 ROS 的双重应激下,酵母细胞活性进一步下降[87]。转录因子 YAP1可促使酵母产生特定抗氧化剂,保护细胞免受 ROS 毒性影响,并提高其对 ROS 的耐受性[88-89]。因此,表达 YBP1 可帮助酵母消耗生长过程中产生的 ROS,并通过表达与细胞壁完整性相关的基因SSD1 提高酵母对乙醇的耐受性。经过上述代谢工程改造后的菌株,其 PPD的最终产量达到了(4.25±0.18) g/L[90]。
在MVA生源合成途径中,从acetyl-CoA到2,3-环氧鲨烯的合成需要 10 种酶的依次催化。通过模块化工程策略优化 MVA 途径中的所有相关基因,可促进 acetyl-CoA 的大量积累,使工程菌株中的 PPD 产量达到 8.09 g/L[91]。基于相同的模块化工程策略,通过引入 PPD 后续合成所需的基因(包括 synPgDDS、synPgPPDS、synPgCPR1),并增加 synP‐ gPPDS 的拷贝数,PPD 的产量可以进一步提高至 11.02 g/L[92]。此外,利用酵母细胞中代谢反应常集中于特定细胞器的特点,通过过表达与过氧化物酶体生成相关的基因(PEX34、PEX11 和 ATG3),增加过氧化物酶体的拷贝数和膜面积,将PPD生物合成途径重构于过氧化物酶体中,从而将PPD产量提高了约 78%[93]。除了上述代谢工程方法,还可以通过优化 CYP450 酶的辅因子 NADPH 的供应,改善 CYP450 的催化效率,从而进一步提高 PPD 的产量[94]。
3.1.2 不同 PPD 型稀有人参皂苷的代谢工程研究
通过在高产 PPD 的工程菌株中引入对应的尿苷二磷酸-糖基转移酶(UDP-glycosyltransferases,UGTs),可实现不同PPD型稀有人参皂苷的合成,但在此过程中PPD合成前体的优化必不可少。例如,酿酒酵母中的磷酸酶LPP1负责将FPP转化为法尼醇,其失活可减少FPP的消耗。将密码子优化的来源于三七的 C-K 合酶基因 SynPn3-29 与尿苷二磷酸葡萄糖 (UDPG)合成途径基因整合到高产PPD酵母菌株的 LPP1基因位点,通过破坏LPP1基因失活该合成酶,从而将更多代谢流引导至 C-K 生物合成途径,最终使C-K产量达到1.17 g/L[95]。此外,UDPG供应不足是限制皂苷高产的普遍问题,增加 UDPG 合成是 1 种重要的策略。PGM2和 UGP1分别编码磷酸葡萄糖变位酶2和UTP-葡萄糖-1-磷酸尿苷转移酶1,共同参与UDPG的合成[96]。在高产PPD的菌株中过表达 PGM2和 UGP1,并以甘油和乙醇为碳源,可以使 C-K 产量提高到(1.70±0.16) g/L[97]。除了增强 UDPG 的供应,降低 UDPG 的消耗也可以进一步提高C-K的产量。Wang等[98] 通过删除FKS1、GLC3和 ALG5基因,显著降低UDPG在细胞壁合成、糖原代谢和蛋白质糖基化中的消耗,优化了 UDPG 的代谢流向 C-K 的糖基化途径,使 C-K 产量从 3.98 g/L 提高至 5.74 g/L。此外,细胞器分区化策略也可提高 C-K的产量。Shi等[99] 利用酵母PLN1蛋白将原定位于内质网的 PPDS 酶靶向到脂质体,将中间体达玛烷二醇到 PPD 的转化效率从 17.4% 提高至 86%;然后通过调控脂质体的体积和形态,优化中间体的储存和酶促反应环境,使 C-K 的产量在 5 L 发酵罐中达到5 g/L,是传统内质网策略的5倍。
通过在产 PPD 的酿酒酵母中引入糖基转移酶 UGTPg45,可以将 PPD 的 C3 位羟基进行糖基化而获得稀有人参皂苷 Rh2。然而,由于 UGTPg45催化效率较低,Rh2 的产量仅为 1.45 μmol/g(细胞干质量,DCW)[100]。随后,通过挖掘三七转录组数据库,发现了 1种催化效率更高的糖基转移酶 UGTPn17。将其引入PPD生产菌株,并以乙醇为发酵碳源,Rh2 的产量提升至354.69 mg/L[101]。一些研究者发现,来自酿酒酵母的糖基转移酶 UGT51 可催化 PPD 转化为 Rh2,但野生型的菌株转化效率较低。利用基于结构信息的半理性蛋白工程策略,获得了突变体 M7-1,其对 PPD 的转化率达到 100%。引入突变酶后,通过减少 Rh2 降解并增强 UDPG 前体供应,使 Rh2 的产量从 0.0032 mg/g DCW 提高到 0.39 mg/g DCW。进一步通过基因拷贝数增加、启动子工程和代谢工程优化,最终将 Rh2 的产量提高至 2.25 g/L,较初始菌株提升了900倍以上[102-103]。
稀有人参皂苷 Rh2 的 C3 位葡萄糖链可通过进一步的糖基化修饰生成另一种稀有人参皂苷 Rg3。例如,在产 PPD 的酿酒酵母菌株基因组中整合 PgUGT74AE2 和 PgUGT94Q2 两种 UGT 基因,可实现 Rg3 的生产,产量约为 1.3 mg/L[104]。通过人参转录组数据库,鉴定出 UGTPg45 基因,将其整合到酿酒酵母底盘后生成 Rg3 的产量为 3.49 μmol/g DCW[100]。然而,UGT 催化效率低下是限制 Rg3 产量的主要原因。从三七的基因组和转录组数据中筛选得到一种催化效率较高的 UGT PnUGT33。将该酶引入酿酒酵母,并结合 UDPG 合成相关基因的表达,使Rg3的产量显著提高至51 mg/L[105]。
PPD 的 C3 和 C20 位羟基可通过糖基化修饰生成稀有人参皂苷 F2。通过 CRISPR/Cas9 系统将 DDS、PPDS、PgUGT74AE2、UGTPg1 和 AtCPR2 整合到酿酒酵母基因组中,成功实现了 F2 的生物合成,但初始产量较低,仅为1.2 mg/L。进一步研究发现,敲除HXK2基因可以增强糖酵解向MVA途径的代谢流量,从而促进外源三萜类化合物的生物合成;过表达转录激活因子 HAC1 可以提高外源酶的表达水平,使F2的产量达到原始菌株的1.9倍;优化 CYP450 还原酶 CPR 的表达,提升了达玛烷二醇到 PPD 的转化效率,突破 F2 合成的限速步骤;然后敲低广谱葡萄糖苷酶EGH1的表达,减少了其对F2的 C3 糖基的水解作用。通过这些代谢工程策略相结合,将F2的最终产量提高至21.0 mg/L[106]。
3.2 PPT型稀有人参皂苷的代谢工程研究进展
PPD在 CYP716A53v2的催化下可合成 PPT,随后通过 UGTs进行糖基化修饰以合成人参皂苷 PPT 型皂苷。通过在酿酒酵母中异源表达 DDS、 CYP716A47、CYP716A53v2和AtCPR1,可实现PPT 的生产[107]。进一步的蛋白质工程策略和半理性设计显著提高了CYP716A53v2的特异性和催化活性。在高产 PPD的酵母底盘菌株中引入该酶,使 PPT的产量超过 5.0 g/L[108]。Wei 等[109] 通过对人参转录组文库中潜在的 UGTs 进行筛选,挖掘出两个能够参与 PPT 型稀有皂苷生物合成的 UGTs——UGTPg1 和 UGTPg100。将 UGTPg1 引入产 PPT 的酵母菌株,可对PPT的C20位羟基进行糖基化修饰,生成稀有人参皂苷F1;而UGTPg100可对PPT的C6位羟基进行糖基化修饰,生成稀有人参皂苷 Rh1。为进一步提升F1和Rh1的产量,作者通过同源模拟技术和点突变验证发现了影响这两种酶催化活性的关键氨基酸残基,并对其进行了定向进化,最终将人参皂苷 F1 和 Rh1 的产量分别提高到 42.1 mg/L 和 92.8 mg/L 的规模。以 UDP-鼠李糖为糖基供体,通过对人参皂苷 Rh1 的 C6 位葡萄糖链进行进一步糖基化修饰,可生成另一种PPT型稀有人参皂苷Rg2。 Li 等[110] 在酵母菌株中引入了 PgDDS、CYP716A47、 CYP716A53v2和 PgUGT71A54,以及从人参转录组数据中鉴定的鼠李糖基转移酶基因PgUGT94,通过引入来源于拟南芥的 AtRHM2,实现了将 UDPG 转化为 UDP-鼠李糖的生物合成路径,最终在酵母中重构了Rg2的生物合成途径。为了进一步提高Rg2 的产量,研究者对 synPgUGT94 进行了关键位点的氨基酸突变,并在酵母菌株中引入了突变体,同时增加了 PgUGT71A54 的基因拷贝数。在代谢工程策略的辅助发酵中,Rg2的最终产量达到1.3 g/L。
利用代谢工程的手段制备稀有人参皂苷的核心是能够在皂苷骨架上不同位点加上特定种类和数量的糖基。因此,挖掘与稀有人参皂苷相关的UGT基因并提高其催化效率是提升微生物细胞工厂生产能力的关键。目前大多数应用的UGTs来自人参属植物,但其他植物和微生物中也存在大量未被开发的 UGT 基因。通过高通量测序技术和生物信息学深入分析植物和微生物的基因组、转录组和蛋白质组数据,有望发现更多具有潜力的UGTs。基于定量质谱和荧光的高通量酶活性检测技术,以及其他高通量筛选方法,可快速检测酶活性并高效筛选优质 UGTs。此外,借助人工智能技术,对稀有人参皂苷 UGTs的晶体结构进行同源建模和活性口袋分析,结合合理设计和结构修饰,可显著提高其催化效率。
4 小结与展望
当前酶催化和代谢工程的研究进展为稀有人参皂苷的高效工业化生产提供了重要的参数和技术支持,但目前仍存在一些需要进一步解决的问题。从酶催化角度而言,面临的主要挑战包括:糖苷酶对糖基位置的特异性和底物适用性不足,导致副产物生成或转化率低,同时部分关键糖基位点的水解难度较大进一步降低了效率;某些糖苷酶在高温、高 pH等工业化条件下稳定性不足,且酶的高成本和难以回收限制了其大规模应用。此外,用于转化的主流人参皂苷前体来源有限且提取成本高,而稀有人参皂苷的结构与药理活性关系研究尚不充分,也为工艺优化带来理论上的局限性。代谢工程方面存在的问题主要有:底物供应不足是高效生产的重要瓶颈,需要通过增强供应和减少消耗来提升产量;代谢通量的不合理分配会导致资源浪费并阻碍目标产物的积累;合成路径涉及多步酶促反应,路径优化不足和工程菌株设计的局限性进一步限制了生产效率。未来的研究方向应集中在以下几方面:开发更多具有高特异性和高转化效率的工程酶,进一步提升稀有人参皂苷的产量与纯度;应用基因工程和蛋白质工程技术优化糖苷酶的结构与功能,以满足不同稀有人参皂苷的产业化需求;利用合成生物学手段构建更加精准的代谢调控网络; 通过细胞器分区化和动态代谢流量控制优化代谢通路;进一步开发智能化发酵技术以实现过程自动化和生产高效化。通过这些改进,不仅能实现稀有人参皂苷的高效绿色生产,还可为其他复杂天然产物的生物合成提供技术借鉴。






